1、PCoA的作图主要分为三个步骤:选择特定的相似性距离并计算距离矩阵。距离的选择可以有Bray-curits、Unifrac等,不同的距离有不同的作用和意义(具体可以参考 微生物β多样性常用计算方法比较)。

2、相似性距离可以利用R的GUniFrac和vegan等包计算,也可以利用QIIME计算。进行PCoA分析,也就是利用表征分析选择最能表示样本距离的坐标轴。这个可以利用R的ape包的pcoa()命令完成。

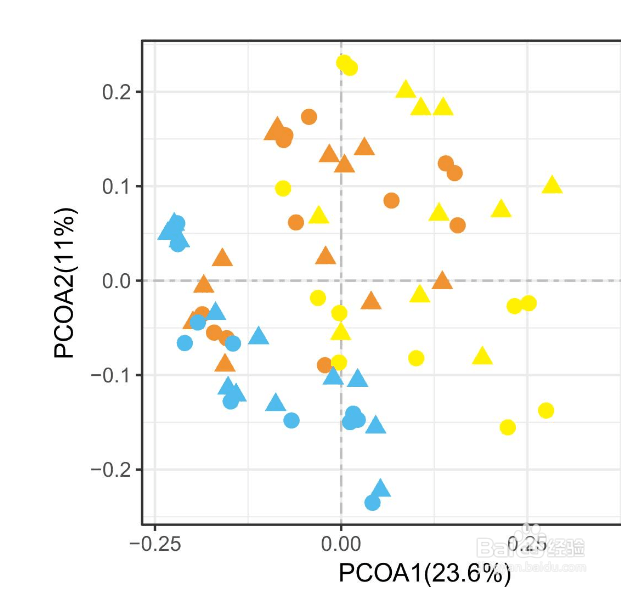
3、PCoA图形展示。图形可以用ordiplot()命令展示,但如果需要比较美观的图形,建议用ggplot来画。

4、###导入需要的R包library(GUniFrac)#用于计算Unifrac距离library(ape) # 用于pcoa分析library(ggplot2) #用于画图##读文件Otu_tab <- read.table('otu_table',row.names="">Tree <- read.tree('muscle.align.tree.nhx')="">#输入OTU表格Otu_tab <->Otu_tab_rff <->unifracs <- gunifrac(otu_tab_rff,tree,alpha="c(0," 0.5,="">du <- unifracs$unifracs[,="" ,="" 'd_uw']=""># 计算Unweighted UniFrac距离

5、Group <- c('a'挢旗扦渌;,'b','c')="">#按照目的输入样本shape <- c('a'="16,'B'" =17,'c'="16)">#定义点形状color <- c('a'='#CCFF33' ,'b'='#CCFF33' ,'c'='#CCFF33' )="">#定义点颜色PCOA <- pcoa(du,="" correction='none' ,="" rn="NULL)">#利用PCOA()指令做pcoa分析